胃癌是全球最常见的恶性肿瘤之一,在癌症死亡原因中高居前列1, 2。在中国,胃癌的发病率和死亡率均位于恶性肿瘤的第二位3。研究显示,胃癌病人的5年生存率约为25%;发生转移的胃癌病人约占胃癌患者的40%,其5年生存率仅为5%4, 5。然而,当前人们对胃癌转移的生物学过程及其分子机理仍知之甚少。因此,深入探究胃癌转移的分子机制及寻找有效的胃癌治疗手段具有重要的意义。
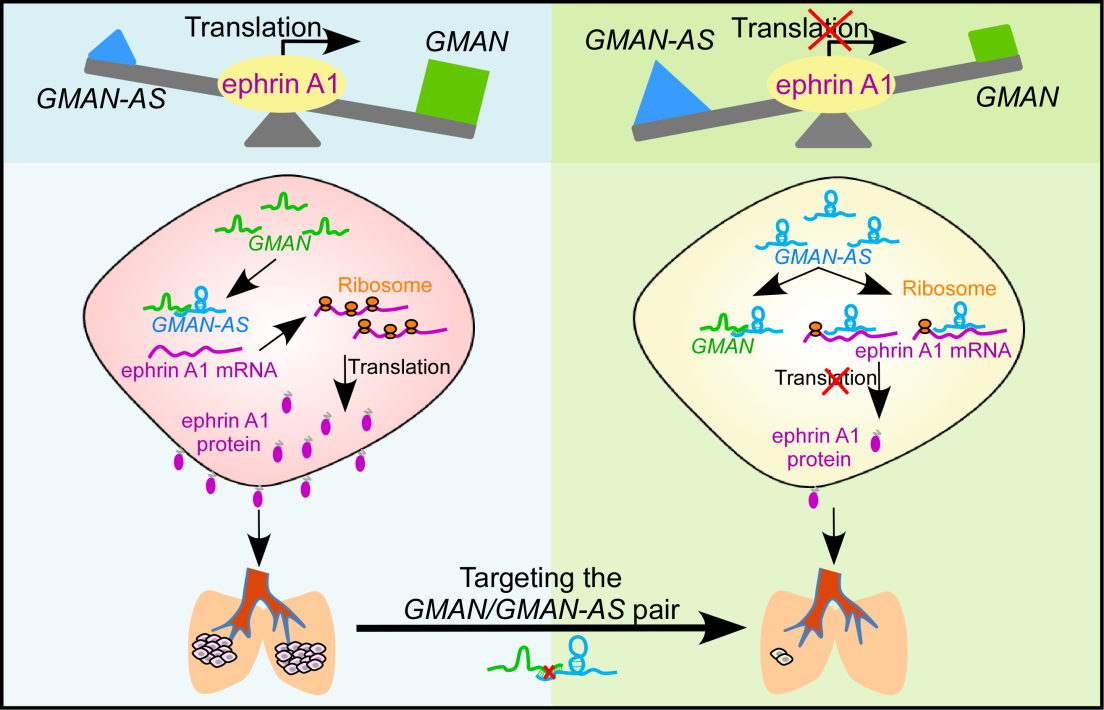
近日,浙江大学基础医学院周天华教授研究小组在Gastroenterology(胃肠病学)杂志(影响因子20.7)上发表题为“Long Non-coding RNA GMAN, Upregulated in Gastric Cancer Tissues, is Associated with Metastases in Patients and Promotes Translation of Ephrin A1 by Competitively Binding GMAN-AS”的研究成果,该研究筛选到一个与胃癌转移及患者预后密切相关的长链非编码RNA(long non-coding RNA, lncRNA),并命名为GMAN (gastric cancer metastasis associated long noncoding RNA)。进一步研究发现GMAN是一个正义lncRNA,可竞争性地与其对应的反义lncRNA(GMAN-AS)相互作用,调控靶基因ephrin A1的翻译过程,显示三个碱基互补RNA分子间的相互作用可调节靶基因的翻译过程,揭示细胞内存在一类由正义lncRNA介导的新型遗传信息传递方式。本研究发现了GMAN/GMAN-AS/ephrin A1调控轴在胃癌转移过程中发挥重要作用,可为胃癌转移治疗提供潜在的新策略。
近年来的研究显示,只有不到2%的人类基因组DNA具有编码蛋白的能力,至少75%的基因组DNA可转录为非编码RNA,暗示着生物体内可能存在大量的非编码RNA6-8。根据非编码RNA分子的大小可将其分为:长度小于200个核苷酸的非编码小RNA和长度大于200个核苷酸的长链非编码RNA (lncRNA)。一般认为lncRNA主要由RNA聚合酶II (Pol II)转录生成,常具有甲基鸟苷帽子 (m7GPPPN)和多聚腺苷酸尾 (polyA)结构9-11。越来越多的证据显示,lncRNA可通过“向导(guide)”、“诱饵(decoy)”、“支架(scaffold)”或“信号(signal)”等方式,在表观遗传修饰、基因转录、转录后调控、翻译等过程中发挥重要的调节作用,进而影响多种细胞生物学行为,其表达或功能异常与多种疾病的发生发展密切相关12-15。但是,关于lncRNA在胃癌发生发展过程中的具体作用机制目前还知之甚少。
周天华课题组发现了一个功能未知的lncRNA GMAN不仅在胃癌组织中高表达,还与胃癌病人的转移和不良预后显著相关。为了解GMAN在胃癌发生发展中的作用及分子机制,他们利用RNA干扰(siRNA和shRNA)、基因组编辑(CRISPR/Cas9系统)及外源表达基因等手段,在胃癌细胞中改变GMAN的表达,结果显示GMAN能够显著促进胃癌细胞的侵袭和转移。通过基因克隆、生物信息学分析、核糖体谱实验等方法,发现GMAN是位于ephrin A1基因簇中的正义lncRNA,能通过增强ephrin A1 mRNA 的翻译效率,促进胃癌细胞的侵袭和转移。
进一步的机制研究发现,在GMAN的反义链上存在一个未被报道的lncRNA GMAN-AS (GMAN antisense RNA ),其与ephrin A1也存在部分序列的互补配对。GMAN-AS可直接与ephrin A1 mRNA结合,抑制ephrin A1的翻译。RNA pull-down等实验显示GMAN、GMAN-AS和ephrin A1间存在相互作用,竞争性调控ephrin A1 mRNA的翻译进而影响胃癌的转移。当细胞内GMAN-AS含量相对比较高时, GMAN-AS可与ephrin A1 mRNA结合,抑制ephrin A1的翻译和胃癌细胞的转移;当细胞中GMAN含量相对比较高时,GMAN可与GMAN-AS结合,导致GMAN-AS与ephrin-A1 mRNA的结合减少,促进ephrin A1的翻译和胃癌细胞的转移。这项研究还发现,临床胃癌组织中GMAN和GMAN-AS的比例不仅与ephrin A1蛋白水平正相关,还显著影响胃癌病人的预后。
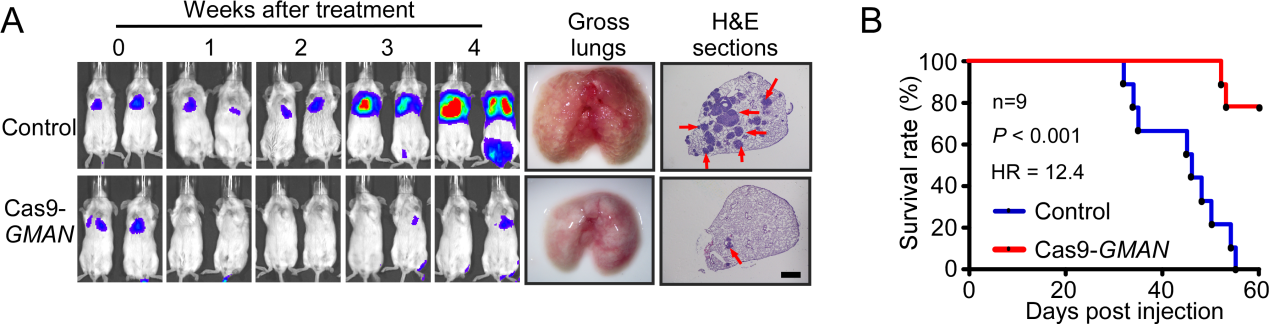
同时,该研究还利用靶向GMAN的CRISPR/Cas9基因组编辑技术,在小鼠体内进行胃癌转移治疗实验,结果显示靶向GMAN的处理组能够显著的抑制胃癌细胞的转移,延长小鼠的生存时间。这些结果提示,靶向GMAN基因的CRISPR/Cas9基因组编辑技术具有一定的潜在临床价值。
综上所述,该研究发现了一种lncRNA调控基因翻译表达的新模式,揭示了lncRNA调控胃癌转移的新机制,探索了基于CRISPR/Cas9技术靶向lncRNA的策略控制肿瘤转移的潜在临床应用前景。
据悉,此项工作主要是由周天华课题组与美国普林斯顿大学康毅滨教授合作完成,得到了浙江大学姒健敏、刘伟、金勇丰等教授的大力帮助。副教授卓巍、博士生刘易曼是论文的共同第一作者。研究项目得到了国家自然科学基金重点国际合作项目和重大研究计划重点支持项目等的资助。
参考文献
1. Torre LA., et al., Global Cancer Statistics, 2012. Ca-a Cancer Journal for Clinicians 2015;65: 87-108.
2. Hayakawa Y., et al., Oesophageal adenocarcinoma and gastric cancer: should we mind the gap? Nature Reviews Cancer 2016;16: 305-318.
3. Chen, W., et al., Cancer statistics in China, 2015. CA: a cancer journal for clinicians, 2016. 66(2):115-132.
4. Bernards, N., et al., No improvement in median survival for patients with metastatic gastric cancer despite increased use of chemotherapy. Annals of oncology, 2013. 24(12): 3056-3060.
5. De Angelis, R., et al., Cancer survival in Europe 1999–2007 by country and age: results of EUROCARE-5—a population-based study. The lancet oncology, 2014. 15(1): 23-34.
6. Djebali, S., et al., Landscape of transcription in human cells. Nature, 2012. 489(7414): 101-108.
7. Gutschner, T. and S. Diederichs, The hallmarks of cancer: a long non-coding RNA point of view. RNA biology, 2012. 9(6): 703-719.
8. Iyer, M.K., et al., The landscape of long noncoding RNAs in the human transcriptome. Nature genetics, 2015. 47(3): 199-208.
9. Chen, L.-L. and G.G. Carmichael, Decoding the function of nuclear long non-coding RNAs. Current opinion in cell biology, 2010. 22(3): 357-364.
10. Laurent, G.S., C. Wahlestedt, and P. Kapranov, The Landscape of long noncoding RNA classification. Trends in Genetics, 2015. 31(5): 239-251.
11. Quinn, J.J. and H.Y. Chang, Unique features of long non-coding RNA biogenesis and function. Nature Reviews Genetics, 2016. 17(1): 47-62.
12. Wang, K.C. and H.Y. Chang, Molecular mechanisms of long noncoding RNAs. Molecular cell, 2011. 43(6): 904-914.
13. Fang, Y. and M.J. Fullwood, Roles, functions, and mechanisms of long non-coding RNAs in cancer. Genomics, proteomics & bioinformatics, 2016. 14(1): 42-54.
14. Mercer, T.R., M.E. Dinger, and J.S. Mattick, Long non-coding RNAs: insights into functions. Nature Reviews Genetics, 2009. 10(3): 155-159.
15. Cesana, M., et al., A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA. Cell, 2011. 147(2): 358-369.











