普通脆性位点
人类细胞的每一次增殖和分裂,都伴随着全基因组DNA的精确复制。然而,在快速分裂的肿瘤细胞中,DNA复制常常会发生问题,导致染色体产生缺口或断裂。半个世纪前,人们就观察到肿瘤细胞的染色体容易断裂,且常常发生在一些固定的保守区域,被称为普通脆性位点(Common Fragile Sites)。在复制压力存在情况下,普通脆性位点处易发生染色体重排和基因拷贝数变异,造成基因组不稳定。脆性位点的“脆性”导致其关联基因易发生突变,因此一直是肿瘤生物学领域的研究热点问题。最为经典的脆性位点FRA3B和FRA16D分别包含抑癌基因FHIT和WWOX,其缺失与肿瘤的发生发展和临床预后差密切相关。普通脆性位点还是病毒癌基因整合与神经疾病相关基因的热点位置,比如FRA7J处的AUTS2和IMMP2L基因突变是导致自闭症的重要原因,而FRA16D处的WWOX基因最近被发现是阿尔兹海默症的敏感基因。虽然普通脆性位点非常不稳定,但是却存在跨物种保守性,这种矛盾关系暗示其可能具有更深层的生物学意义,例如为进化提供遗传突变原料,从而在生物进化中得以保留。
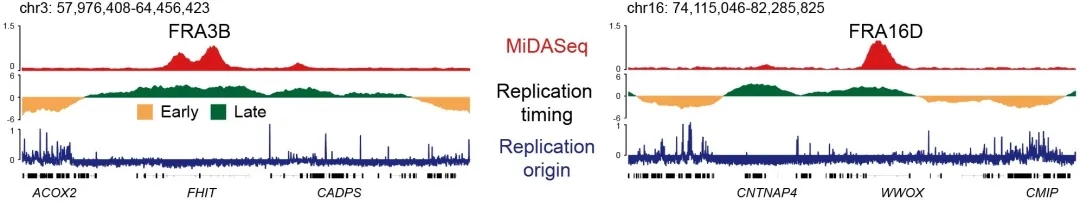
经典脆性位点FRA3B和FRA16D的复制特征
近日,应颂敏、沈华浩教授团队与日内瓦大学Thanos D. Halazonetis教授团队在国际权威学术期刊Cell Research“背靠背”发表了题为Genome-wide High Resolution Mapping of Mitotic DNA Synthesis Sites and Common Fragile Sites by Direct Sequencing的研究论文,首次揭示了普通脆性位点的全基因组序列图谱,为研究其生物学意义和相关疾病治疗靶点提供了重要依据。浙江大学医学院/附属第二医院呼吸与危重症医学科应颂敏教授和沈华浩教授为该论文的共同通讯作者。近年来,该研究团队连续发表系列工作,系统性揭示了肿瘤细胞在DNA复制压力存在情况下的损伤修复机制(Nature,2015;Nature Cell Biology,2013;Molecular Cell,2020),为研究普通脆性位点的基因组定位奠定了基础。
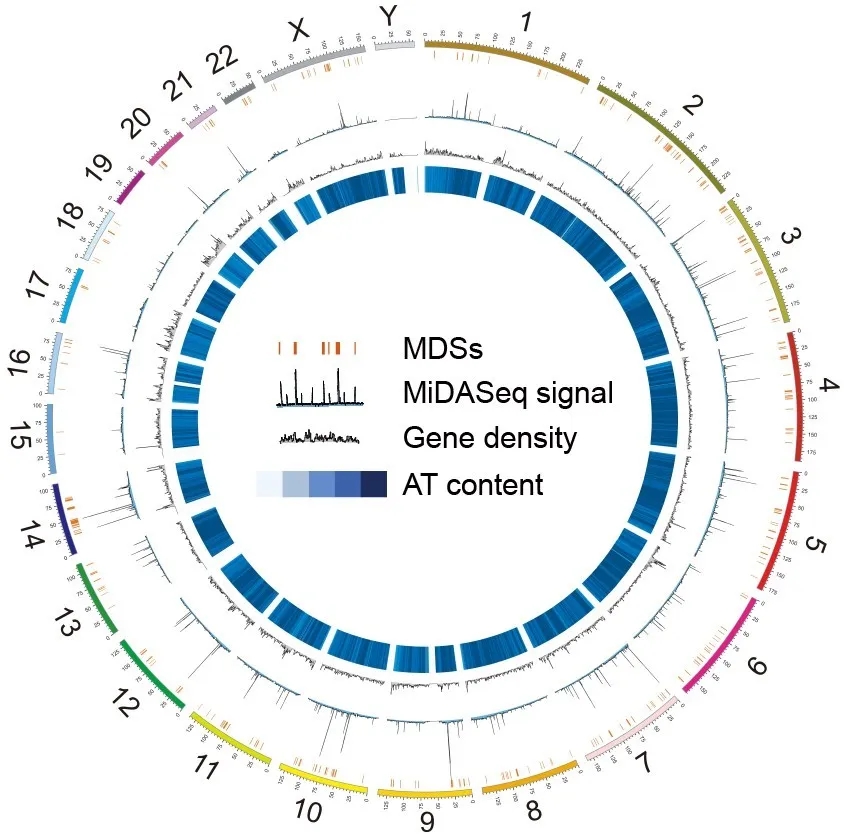
普通脆性位点在全基因组的分布情况
“脆性”背后的序列特性
论文第一作者、浙江大学基础医学院2018级博士研究生季芳指出,尽管人们对于脆性位点的研究已有将近半个世纪,但是由于其数量众多且缺乏特异的富集技术导致绝大部分脆性位点的DNA序列仍不明确,目前的分辨率仍停留在MB级别。因此研究其“脆性”背后的序列特性和分子机制仍充满挑战。在本研究中,研究者利用普通脆性位点会在有丝分裂期进行DNA合成这一生物学特征,对其进行富集和高通量测序,从而获得了普通脆性位点的精确坐标。
值得一提的是,多位来自浙江大学医学院临床医学八年制(竺可桢学院巴德年班)的本科同学参与了这项研究。该论文的共同第一作者、2017级本科生潘晟指出,通过序列分析和表观遗传分析,他们发现普通脆性位点富含AT序列、难以复制以及转录抑制的特征,其倾向于在细胞周期较晚的阶段进行复制,而且区域中缺乏复制起始位点。这些分析结果验证了传统的认知和猜想。更重要的是,他们发现超大基因的长转录本转录和复制时间的滞后是导致普通脆性位点脆性的决定性因素。此外,该工作还通过物种比对,发现人的普通脆性位点涵盖绝大部分小鼠的双链断裂热点区域,再次证实了普通脆性位点的物种保守性。










